6165cc金沙总站检测中心院讯(通讯员:田圣文)近日,生物信息领域国际顶级期刊IEEE Journal of Biomedical and Health Informatics接收我院学术论文(Sheng-Wen Tian, Jian-Cheng Ni, Yu-Tian Wang, Chun-Hou Zheng and Cun-Mei Ji. scGCC: Graph Contrastive Clustering with Neighborhood Augmentations for scRNA-seq Data Analysis, 2023)。该论文以6165cc金沙总站检测中心为第一完成单位,2021级研究生田圣文为论文第一作者,倪建成教授和嵇存美副教授为论文共同通讯作者。IEEE Journal of Biomedical and Health Informatics是中科院1区TOP期刊,最新影响因子为7.7。
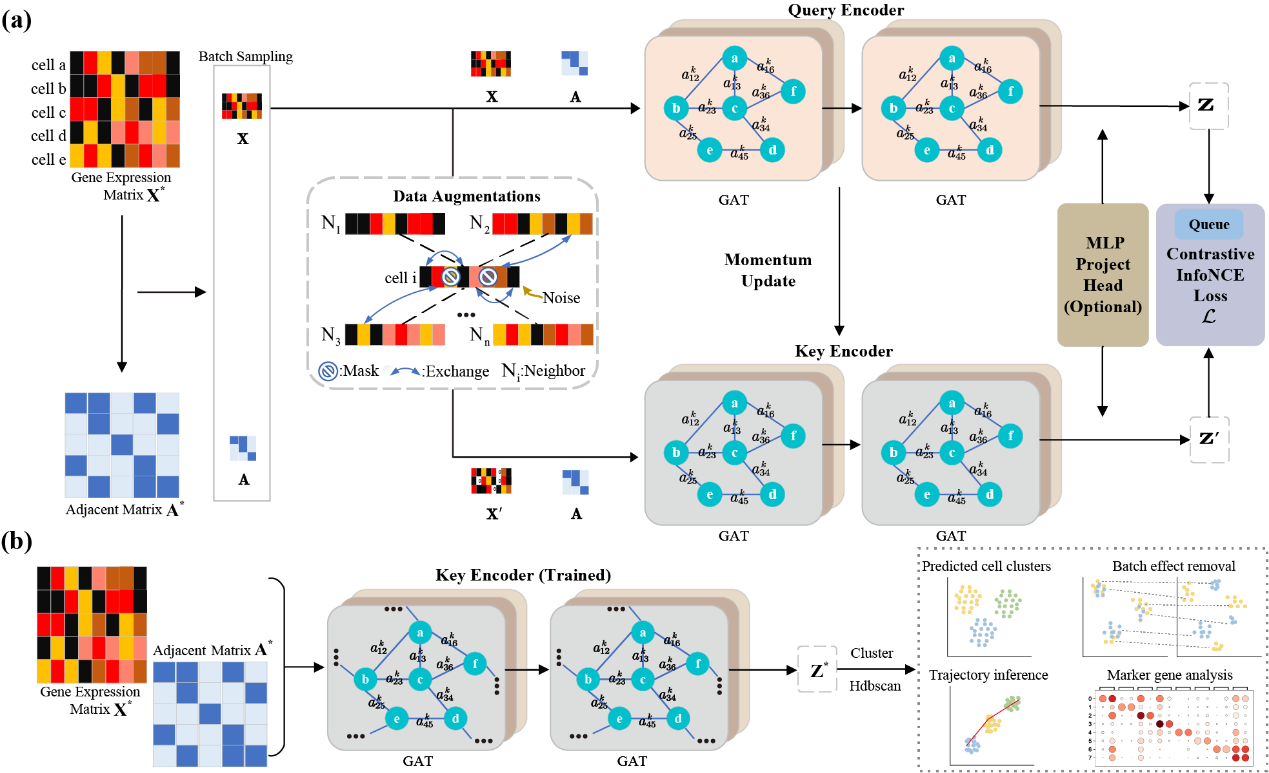
在scRNA-seq数据分析中,细胞聚类是下游分析的关键步骤,因为它使我们能够识别细胞类型并发现新的细胞亚型。然而,scRNA-seq数据的特性,如高维度、稀疏性、dropout事件和批次效应,为聚类分析带来了显著的计算挑战。在本研究中,我们提出了scGCC,一种新颖的图自监督对比学习模型,以解决scRNA-seq数据分析中面临的挑战。scGCC包括两个主要组件:表示学习模块和聚类模块。首先,将scRNA-seq数据输入表示学习模块进行训练,然后通过聚类模块进行数据分类。scGCC可以学习低维去噪嵌入,这对我们的聚类任务非常有优势。我们引入了图注意力网络(GAT)进行细胞表示学习,从而实现更好的特征提取和提高聚类准确性。此外,我们提出了五种数据增强方法,通过增加数据多样性和减少过拟合来提高聚类性能。这些方法增强了聚类结果的鲁棒性。我们对14个真实世界数据集进行了实验研究,结果表明我们的模型在准确性和鲁棒性方面表现出色。我们还进行了下游任务,包括批次效应去除、轨迹推断和标记基因分析,以验证我们模型的生物学有效性。
全文链接:https://doi.org/10.1109/JBHI.2023.3319551
该研究得到了国家自然科学基金的资助支持。